最近、Dockerを使ってPsi4とRDKitが使える環境を構築したのでやり方をまとめておこうと思います。
Dockerを利用すると以下のようなメリットがあります。
- 再現性:
Dockerfileとdocker-compose.ymlを共有することにより、第三者も同一の実行環境を正確に再現可能です。共同研究や成果物の公開において有効です。 - 環境の独立性: ホストOSの環境を汚染することなく、プロジェクトごとに完全に独立したライブラリ環境を維持できます。
▼ 筆者のケモインフォマティクスおすすめ本
事前準備
Docker Desktop がローカルマシンにインストール済みであること。
プロジェクト雛形を作成
chem_psi4_env/
├─ docker-compose.yml
├─ Dockerfile
├─ environment.yml
└─ src/
└─ psi4_test.ipynb
srcディレクトリは、Jupyter Notebookファイル(.ipynb)など、実際の分析コードやデータを格納する作業用スペースとして使います。
設定ファイルの準備
各設定ファイルの役割と内容を以下に示します。
environment.yml
Condaの仮想環境を定義するファイルです。インストールするライブラリとそのバージョンを指定します。environment.yml に以下の内容をコピーします。
name: chemoinf
channels:
- conda-forge
dependencies:
- python=3.11
- psi4
- rdkit
- numpy
- pandas
- matplotlib
- seaborn
- scikit-learn
- plotly
- jupyterlab
- ipykernel
- lightgbm
- xgboost
- py3dmol
- pubchempy
- mordred
Dockerfile
Dockerイメージを構築するための手順を記述したファイルです。minicondaの公式イメージをベースとして、environment.ymlに基づいたConda環境を構築します。
# ベースイメージとしてcontinuumio/miniconda3:latestを使用
FROM continuumio/miniconda3:latest
# 作業ディレクトリを設定
WORKDIR /workspace
# システムライブラリのインストール
RUN apt-get update && apt-get install -y \
libxrender1 libxext6 libsm6 \
&& rm -rf /var/lib/apt/lists/*
# Conda環境定義ファイルをコンテナにコピー
COPY environment.yml .
# environment.ymlからConda環境を作成し、デフォルトで有効化
RUN conda env create -f environment.yml && \
echo "conda activate chemoinf" >> ~/.bashrc
# PATH環境変数を設定
ENV PATH /opt/conda/envs/chemoinf/bin:$PATH
# 作業ディレクトリをsrcに変更
WORKDIR /workspace/src
# コンテナ起動時のデフォルトコマンドを設定
CMD ["bash", "-c", "source activate chemoinf && jupyter lab --ip=0.0.0.0 --allow-root --no-browser --NotebookApp.token="]この設定により、コンテナ起動時にchemoinf環境が自動で有効化され、Jupyter Labが起動します。
docker-compose.yml
Dockerコンテナの構成と起動設定を定義するファイルです。これにより、単一のコマンドでサービスを管理できます。
version: "3.3"
services:
python:
build: .
container_name: chemoinf_psi4
volumes:
- ./src:/workspace/src
ports:
- "8888:8888"
environment:
- JUPYTER_ENABLE_LAB=yesbuild: .: カレントディレクトリのDockerfileを使用してイメージをビルドします。volumes: - ./src:/workspace/src: ローカルのsrcディレクトリをコンテナ内の/workspace/srcにマウントし、ファイルの同期を可能にします。ports: - "8888:8888": ローカルホストのポート8888をコンテナのポート8888にフォワーディングします。
コンテナの起動と動作確認
起動コマンド
上記ファイルの準備が完了したら、ターミナルでプロジェクトのルートディレクトリに移動し、以下のコマンドを実行します。
docker-compose up -d --build--buildオプションは初回のみ必須です。2回目以降はイメージがキャッシュされるため、docker-compose up -dのみで起動します。
動作確認
コンテナが正常に起動したら、Webブラウザで http://localhost:8888 にアクセスします。Jupyter Labのインターフェースが表示されます。
ファイルブラウザからsrc/psi4_test.ipynbを開き、以下のコードセルを実行して、ライブラリが正しくインストールされ、動作することを確認します。
水分子の構造最適化のテストコード
import psi4
psi4.set_output_file('h2o_opt.log')
h2o = psi4.geometry('''
0 1
O 0.000000 0.000000 0.113960
H 0.000000 0.748816 -0.455842
H 0.000000 -0.748816 -0.455842
''')
psi4.optimize('hf/sto-3g', molecule=h2o)
print(h2o.save_string_xyz())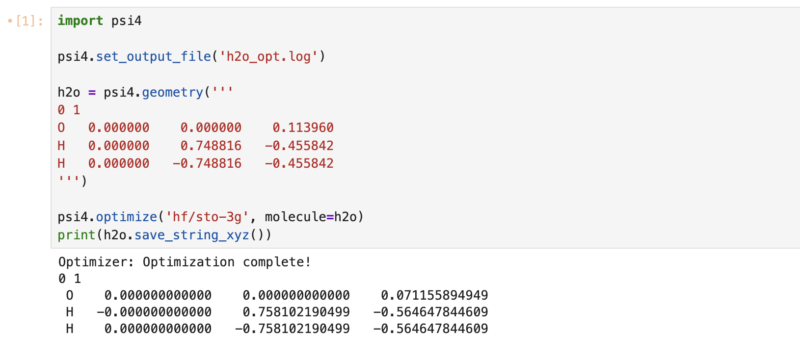
このような出力であればokです。
まとめ
Dockerを利用することで、複雑な依存関係を持つライブラリを含む化学計算・ケモインフォマティクス環境を、容易に構築できます。再度Dockerを使うメリットをまとめておきます。
- 再現性:
Dockerfileとdocker-compose.ymlを共有することにより、第三者も同一の実行環境を正確に再現可能です。共同研究や成果物の公開において有効です。 - 環境の独立性: ホストOSの環境を汚染することなく、プロジェクトごとに完全に独立したライブラリ環境を維持できます。
- セットアップの簡便性:
docker-compose upコマンドを実行するだけで、必要なコンポーネントがすべて導入された環境を利用できます。




コメント