今回はDockerとVS Codeを使って、rdkit・py3Dmol・PubChemPyをインストールした化学でデータサイエンス、ケモインフォマティクスを行うための環境を作成する方法を紹介します。
この記事でわかること
- 再現性の高いケモインフォマティクスの環境構築(必要なライブラリが増えればrequirements.txt に足すだけ)
- rdkit・py3Dmol・PubChemPy を動かせる Dockerfile/docker-compose.yml の構成
- VS Code上で分子を 2D/3D 表示するテストコード(ブラウザからJupyterでも可能)
- そのまま機械学習にも使える環境が作れる
環境構築の方法は以前書いた記事に基づいています。もちろん、本記事を読むだけでも環境構築できます。
本記事の環境構築で使用するツール
環境構築には以下のツールを使用します。
| ツール | ダウンロード |
|---|---|
| Docker Desktop | https://www.docker.com/ |
| Visual Studio Code | https://code.visualstudio.com/ |
ライブラリ
RDKit、Py3Dmol、PubChemPyは、いずれもPythonで化学構造や化学データを扱う際に非常に役立つライブラリです。組み合わせることで以下のような使い方ができます。
PubChemPyを使ってPubChemから関心のある化合物のSMILES文字列を取得する。
↓
取得したSMILES文字列を RDKit で読み込み、分子記述子を計算する。機械学習モデルを構築・予測する。
↓
RDKit で処理した分子構造や、特に注目したい化合物を Py3Dmol を使ってJupyter Notebook上でインタラクティブに3D表示して確認する。
プロジェクトの構造
以下のプロジェクトをご自身のローカル環境に作成します。desktopフォルダなど任意の場所に作成してください。「src」フォルダは後に説明するように仮想環境と同期(マウント)するので、ここにjupyterやpython用のコードを配置し、VSCodeから開いて、docker環境のインタプリタで動かします。
chem_ds_env/
├── Dockerfile
├── docker-compose.yml
├── .dockerignore
├── requirements.txt
└── src/ ← pythonスクリプトを置く場所
フォルダ作成コマンド(コピペ用)
以下のコードをコマンドラインで実行すれば、必要なプロジェクトフォルダが作成されます。
mkdir chem_ds_env && cd $_
mkdir .devcontainer src
touch Dockerfile docker-compose.yml .dockerignore requirements.txt 以下の画像のように実行すればokです。

設定ファイル
各ファイルが作成できたら、以下のコードをそれぞれのファイルにコピペします。
Dockerfile
環境構築に必要なパッケージ、ライブラリのインストールなどを管理するファイルです。
# Python環境を構築
FROM python:3.11-slim
# 作業ディレクトリ
WORKDIR /workspace
# システム依存のインストール
RUN apt-get update && apt-get install -y \
build-essential git \
libxrender1 libxext6 libsm6 \
&& rm -rf /var/lib/apt/lists/*
# 必要なパッケージをインストール
COPY requirements.txt .
RUN pip install --no-cache-dir -r requirements.txt
# 作業ディレクトリに移動
WORKDIR /workspace/src
# Jupyter Labを起動
CMD ["jupyter", "lab", "--ip=0.0.0.0", "--allow-root", "--no-browser", "--NotebookApp.token="]requirements.txt
requirements.txtを使うことで必要なライブラリのインストールをまとめて行えます。
他に必要なライブラリがあれば、こちらに書き足して再度環境構築すれば使えるようになります。
numpy==1.24.3
pandas==2.1.1
matplotlib==3.8.0
seaborn==0.13.0
scikit-learn==1.3.1
plotly==5.17.0
jupyterlab==4.0.7
ipykernel==6.25.2
lightgbm==4.3.0
xgboost==2.0.3
rdkit==2023.09.6
py3Dmol==2.2.0
PubChemPy==1.0.4
mordred==1.2.0今回、RDKitは2023.09.6のバージョンを使用しています。ケモインフォマティクス関連ですと、しれっと、mordredもインストールしてます。簡単な機械学習ができるように、lightgbmとxgboostも入れておきました。
docker-compose.yml
dockerで構築する仮想環境を管理するためのファイルです。
version: "3.3"
services:
python:
build: .
container_name: chemoinf
volumes:
- ./src:/workspace/src
ports:
- "8888:8888"
environment:
- JUPYTER_ENABLE_LAB=yes
.dockerignore
.dockerignoreは.gitignoreのようにdockerでビルドする際に無視するファイルを指定することができます。なくても良いですが、これによって、多少環境構築の時間が短縮されるのに加えて、仮想環境の容量が節約できます。
.git
__pycache__/
.ipynb_checkpoints/
*.py[cod]
*.so
*.dylib
/data/
.vscode/
.DS_Store環境の立ち上げ
コマンドライン上で、作成したプロジェクトのディレクトリ(Dockerfileがある場所)で以下を実行します。
docker compose up -d以下の画像のようにすればokです。
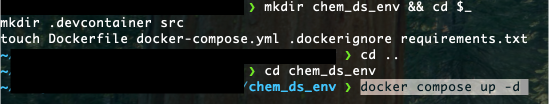
実行すると、いろいろインストールが始まります。ネット環境にもよりますが、5~10分ほどで終わります。最後に、「Container chemoinf Started」と表示されていればokです。(下の画像ではchemoinf_2になってますが気にしないでください。)

ちなみに起動できると「docker ps -a」コマンドで返される「status」が「up」になります。

「docker ps -a」コマンドで確認できる「CONTAINER ID」という文字列を以下のように指定すれば、環境の停止と再起動が行えます。使わないときは停止しておきましょう。
コンテナ(環境)を停止するときは
docker stop "コンテナID"コンテナ(環境)を再起動するときは
docker restart "コンテナID"で行えます。
Jupyterを開く(使いたい人のみでok)
後述するように、本記事ではVSCodeで動かす方法を紹介しますが、「Jupyterでいいよ」って方は、環境が起動していれば、ブラウザから「http://localhost:8888」にアクセスすればJupyterを開けます。
VScodeでdockerの環境に接続する
環境が起動していれば、VSCodeを開いて左下の「> <」を押しましょう。
以下のような選択肢が表示されるので、「実行中のコンテナにアタッチ」を開きます。
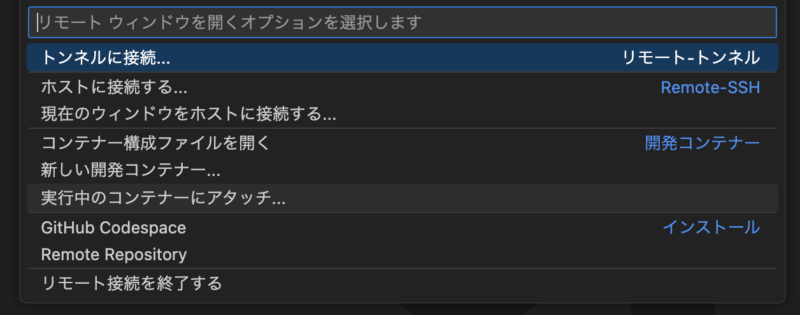
先ほど作成したchemoinfという環境が表示されるので、これをクリックします。下の画像ではchemoinf_2になってますが気にしないでください。)
すると、新しくウィンドウが開かれ、以下のような画面になります。
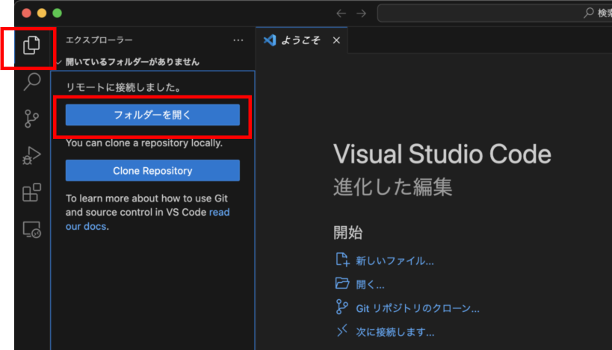
左上の一番上のアイコンをクリックすると、「リモートに接続しました。」と出るので、「フォルダーを開くをクリック」します。
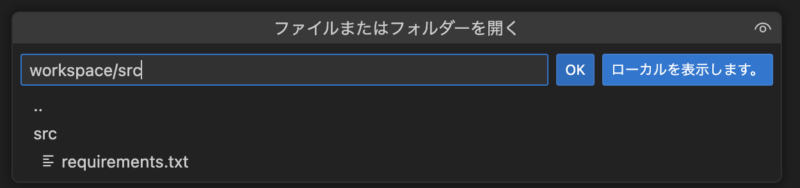
最初「/root」と表示されていますが、これを消して、「workspace/src」と入力すると、マウントされたフォルダが開きます。
py3Dmolで3Dの分子モデルを表示する
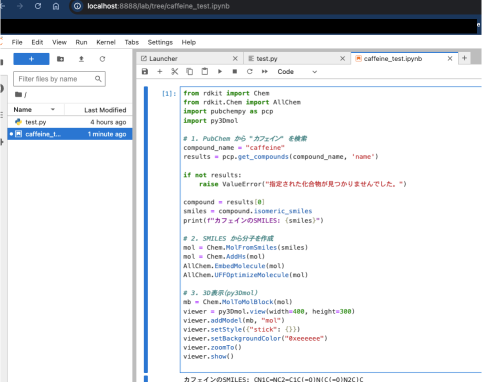
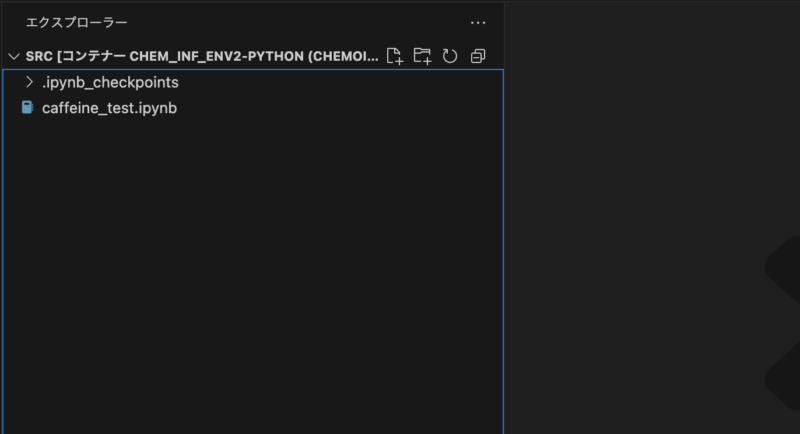
「src」を開いて、「caffeine_test.ipynb」というファイルを作成しましょう。
「caffeine_test.ipynb」に以下のコードを貼り付けます。こちらでpubchempyからカフェインのデータを引っ張ってきてpy3Dmolで表示できます。
from rdkit import Chem
from rdkit.Chem import AllChem
import pubchempy as pcp
import py3Dmol
# 1. PubChem から "カフェイン" を検索
compound_name = "caffeine"
results = pcp.get_compounds(compound_name, 'name')
if not results:
raise ValueError("指定された化合物が見つかりませんでした。")
compound = results[0]
smiles = compound.isomeric_smiles
print(f"カフェインのSMILES: {smiles}")
# 2. SMILES から分子を作成
mol = Chem.MolFromSmiles(smiles)
mol = Chem.AddHs(mol)
AllChem.EmbedMolecule(mol)
AllChem.UFFOptimizeMolecule(mol)
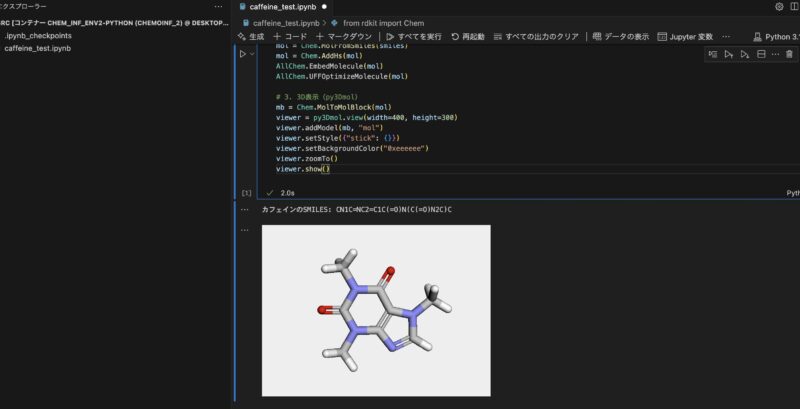
# 3. 3D表示(py3Dmol)
mb = Chem.MolToMolBlock(mol)
viewer = py3Dmol.view(width=400, height=300)
viewer.addModel(mb, "mol")
viewer.setStyle({"stick": {}})
viewer.setBackgroundColor("0xeeeeee")
viewer.zoomTo()
viewer.show()
こちらを実行すると、以下のように分子の3Dグラフが表示されます。無事にVSCode上でJupyterファイルを実行できました!
コードを初めてこの環境で実行すると、インタプリタの選択が表示されるかもしれません。
「Jupyerの拡張機能をインストール」するかとポップが出ると思うので、インストールして大丈夫です。
「カーネルの選択」が表示された場合は、「usr/local/bin/python」を選択しましょう。RDKitで2Dの分子構造を表示する
続いて以下のコードを実行すると、RDKitを使って、2Dのカフェインの構造が表示できます。
from rdkit import Chem
from rdkit.Chem import Draw
mol = Chem.MolFromSmiles("Cn1cnc2c1c(=O)n(c(=O)n2C)C")
img = Draw.MolToImage(mol, size=(300, 300))
img.show() 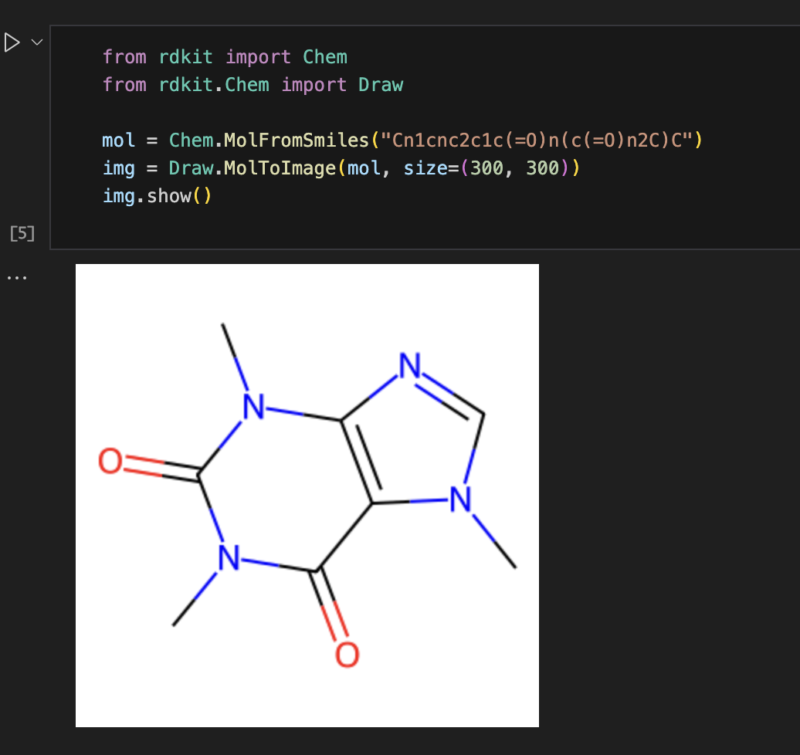
以上のように表示されれば、問題なく環境を動かせています。
おわりに
今回はrdkit・py3Dmol・PubChemPyを使える環境をDockerで作成する方法を紹介しました。
Dockerは環境構築の再現性が高く、手間も少ないので本当におすすめのツールです。
VSCodeで開けると、いろいろ便利ですよね。ぜひ、この環境を機械学習やデータ分析に活用してみてください。
本ブログでも引き続き、RDKitを中心に化学でデータサイエンスを行う記事を書いていくので見てみてください!
最後まで読んでいただきありがとうございました!
P.S. 最近Noteを始めました。化学メーカーの就活のことを発信してるので、興味があればのぞいていただけると嬉しいです。



コメント